Modalanalyse mit Open-Source-Tools – Openmodal
Hallo zusammen!
Im vorherigen Post habe ich einen analytischen Zweimassenschwinger mit dem Software-Tool Easymod identifiziert. Als nächstes schauen wir uns das Bestimmen der modalen Parameter mit Openmodal an. Hierzu muss zunächst die Entwickler-Version unter diesem Link runtergeladen werden. Die auf der Webseite von Openmodal angebotene Alpa release Stellt noch nicht alle Funktionen zur Verfügung. Openmodal ist in Python implementiert, daher müsst ihr zunächst Python auf euren Computer installieren. Am einfachsten ist es, wenn ihr euch Anaconda runterladet und dann eine „Virtuelle Umgebung“ erstellt, in der ihr die benötigten Packages installiert. Wie man ein „Virtuelle Umgebung“ erstellt, ist hier erklärt. Auf der Github Seite von Openmodal ist ebenfall erklärt, welche zusätzlichen Packages ihr benötigt. Openmodal arbeitet wie Easymod mit uff-Dateien. Für dieses Beispiel kann man die von der Easymod Funktion unv58write erstellte uff-Datei verwenden, wenn allerdings mehrere Übertragungsfunktionen in eine uff-Datei gespeichert werden sollen, empfehle ich das von den Entwicklern von Openmodal bereitgestellte Python Package pyuff. Auf der Github Seite von pyuff ist ein Beispiel, wie man mit Hilfe von pyuff eine uff-Datei erstellt. Eine kleine Warnung im Voraus… Openmodal läuft noch nicht sehr stabil, stellt euch also darauf ein, dass es regelmäßig abstürzt. Falls ihr Fragen zur Installation habt, schreibt einfach in die Kommentare oder schaut auf der Github Seite von Openmodal.
Wenn ihr Anaconda installiert, alle Package hinzugefügt,Openmodal und die uff-Datei mit den Übertragungsfunktionen des Zweimassenschwingers geöffnet habt, seht ihr die in Abbildung 1 gezeigt Oberfläche. Nach dem Klicken auf Analyse werden die stabilen Pole (rote Kreuze) angezeigt. Durch Klicken auf eins der Kreuze wird der Pol ausgewählt und die Übertragungsfunktion angezeigt, wie sie nach der Auswahl des Pols aussieht. Dadurch gewinnt man einen guten Eindruck, wie viele Pole notwendig sind, um das Übertragungsverhalten nachzubilden. Bei dem analytischen Zweimassenschwinger ist diese Aufgabe natürlich leicht, weil ja bekannt ist, welche Eigenfrequenzen das System besitzt. In diesem Fall untersuche ich Übertragungsfunktionen H11 und H21. Also die Übertragungsfunktionen beider Massen bei Anregung an der ersten Masse.
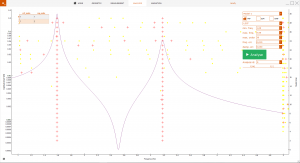
Nach dem Auswählen der Pole müsst ihr die das erzeugt Modell als .mdd Datei abspeichern. Der Export der modalen Parameter ist leider noch nicht automatisiert, daher muss man ein weiteres Python Skript schreiben, um die identifizierten Parameter in eine Excel Datei zu speichern. Der notwendige Code ist hier dargestellt. Das Python Skript muss in der Datei erzeugt werden, in dem sich auch der Openmodal Entwickler Code befindet. Auf der ersten Seite in der Excel Datei sind die Polstellen in der Spalte L abgespeichert. Die Residuen sind auf der zweiten Seite gespeichert. Für den Zweimassenschwinger erhält man vier komplexe Residuen. Für jede Mode und jetzt Masse einen. Die Residuen sind jedoch noch nicht skaliert.
import OpenModal as OM
import pickle
import sys
import pandas as pdsys.modules[‚openModal‘] = OM
file_name = r’C:\Pfad\zu\mdd\Datei\Zweimassenschwinger.mdd‘
f = open(file_name,’rb‘)
data = pickle.load(f)
writer = pd.ExcelWriter(‚Zweimassenschwinger.xlsx‘)
data[0].tables[‚analysis_index‘].astype(str).to_excel(writer, ‚index‘)
data[0].tables[‚analysis_values‘].astype(str).to_excel(writer, ‚values‘)
writer.save()
Die Residuen müssen noch in die Eigenvektoren umgerechnet werden. Am besten ist es auf das Residuum des Knoten der Anregung zu skalieren. Das klingt jetzt wieder sehr theoretisch aber lässt sich sehr leicht am Beispiel des Zweimassenschwingers erklären. Wie oben bereits erwähnt untersuche ich hier die Übertragungsfunktionen bei einer Anregung an der ersten Masse. Also skaliert man jede Spalte der Residuenmatrix auf den ersten Wert in der Spalte. Der Code zur Skalierung der Residuen für das Beispiel des Zweimassenschwingers folgt:
// Residuen Matrix
Aij = [(-4.13206341036e-06+0.0249633328495j), (3.26881850455e-05+0.0124843592375j);
(-5.50210937354e-06+0.0249635526367j),(-3.30042280228e-05-0.0124854226526j)
];
// Skalierung der Residuen
// E sind die komplexen Eigenvektoren des Systems
for ii = 1:size(Aij,2)
E(:,ii) = Aij(:,ii)./sqrt(Aij(1,ii));
end
Mit den Eigenvektoren und den Polstellen können jetzt die Übertragungsfunktionen berechnet werden. Der Scilab-Code zur Berechnung der Übertragungsfunktionen sieht folgendermaßen aus:
function H=modal2frf(f, p, V, indof, outdof)
// f = Frequenzvektor
// p = Polstellen
// V = Eigenvektoren
// indof = Vektor mit dofs (Degree of Freedom) für alle Eingäng
// outdof = Vektor mit dofs für alle Ausgäng
//Umwandeln Frequenz [1/s] in [rad/s]
w = 2*%pi*freq;
//Initialisierung von H
N = length(f)
D = length(outdof)
R = length(indof)
Nm = length(p)
H = zeros(N,D,R)
//For Loop für alle Eingänge
for ii = 1:R
// For Loop für alle Ausgänge
for jj = 1:D
// For Loop für alle Polstellen
for tt = 1:Nm
// Berechnen der (skalierten) Residuen
A = V(indof(ii),tt)*V(outdof(jj),tt);
// Berechnen der Übertragungsfunktion
H(:,jj,ii) = H(:,jj,ii)+A./(%i*w-p(tt))+conj(A)./(%i*w-conj(p(tt)));
end
end
end
endfunction
Wenn ihr die Übertragungsfunktion berechnet habt, könnt ihr euch das Ergebniss plotten lassen. Wie ihr sehen könnt, schafft auch Openmodal das Übertragungsverhalten des Zweimassenschwingers korrekt zu identifizieren. Der Start mit Openmodal ist vielleicht ein bisschen schwieriger aber wenn es einmal läuft, ist es sehr angenehm damit zu arbeiten.
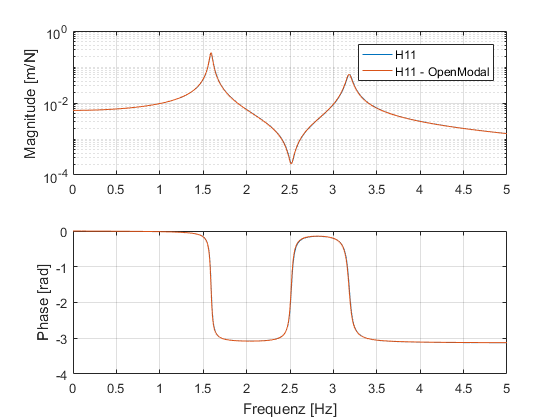
Viel Spaß beim Ausprobieren und ich melde mich bald wieder mit einem Beitrag zu Abravibe.


Schreibe einen Kommentar